米国医学研究者のDr. Pontaです。今回はLinkedOmicsプラットフォームの使い方を簡単に解説します。LinkedOmicsは、がんのマルチオミクスデータを分析するための公開データポータルです。このデータポータルには、TCGAの32がんタイプと臨床プロテオミクス腫瘍分析コンソーシアム(CPTAC)の10がんコホートからのデータが含まれています。では早速みていきましょう。
LinckedOmicsに登録されているデータを確認
LinkedOmicsにアクセスすると、データベース内の利用可能なデータセットの一覧が表示されます。これらのデータセットは、がんの種類や他の疾患に関連するデータなど、さまざまなテーマで分類されています。
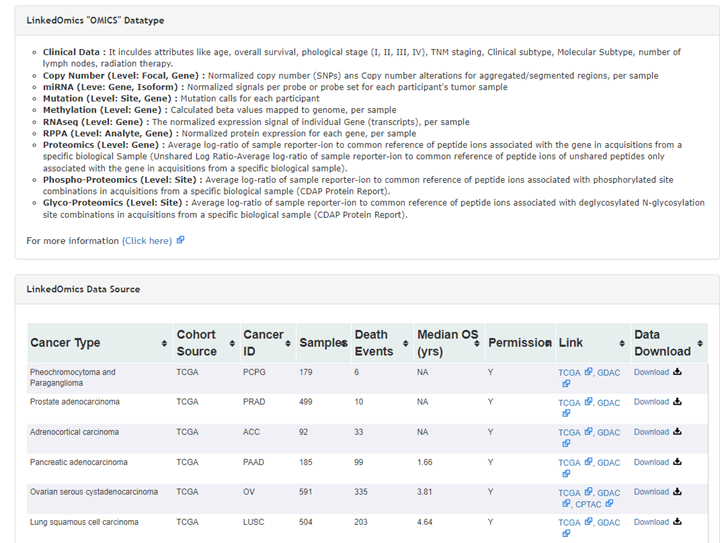
コホートリストのデータダウンロードリンクをクリックすると別のダウンロード専用ページに行けます。
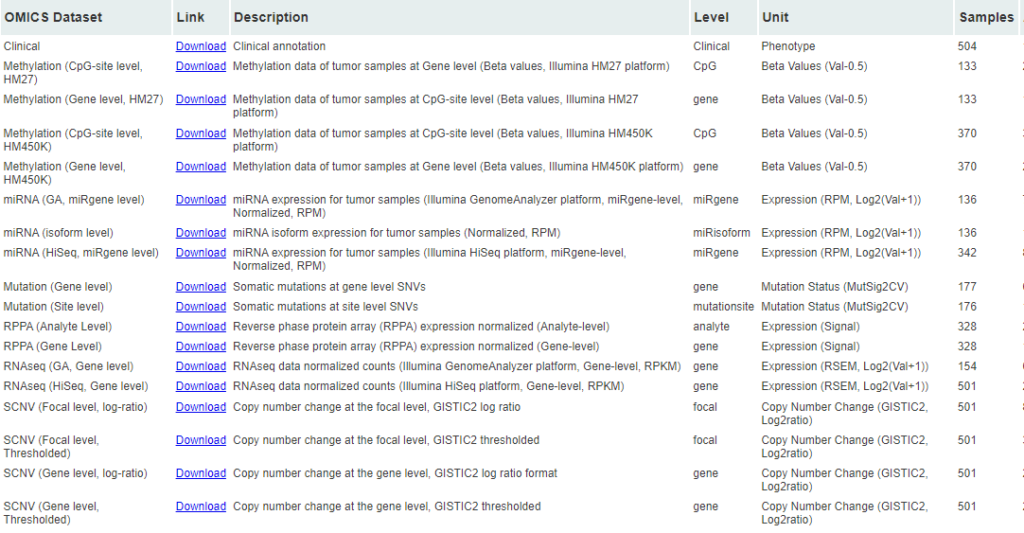
以下は肺扁平上皮癌(TCGA-LUSC)のデータページです。ここからクリックするだけでWESやRNAseqのデータをダウンロードできてします。とても簡単ですね!一度ローカル環境に落としてしまえば、RやPythonでいかようにでも解析できるので、とても便利です。
LinkedOmics上でデータ解析をする
さて次にLinkedOmicsの他重要機能についての紹介です。実はこのウェブサイト上で登録されたデータをcBioPortalのように解析できてしいます。基本的な解析のみですが、必要に応じて使えるようになっておくと便利でしょう。ではさっそく見ていきます。
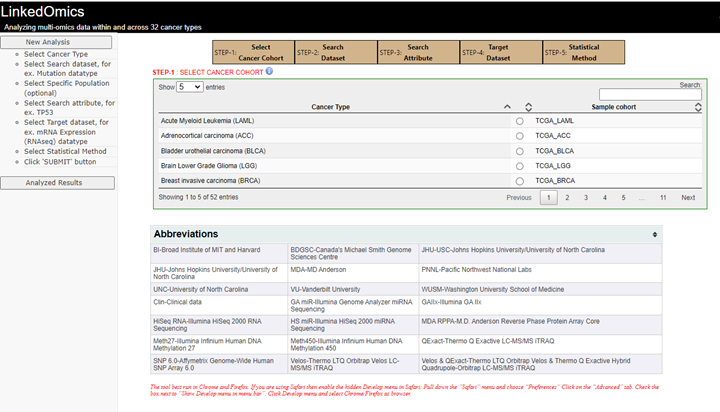
LinkedOmicsログイン
データセットを解析し、プラットフォーム上に保存するにはユーザー登録が必要ですが、そうでない場合はゲストログインでも構いません。ログインすると以下の画面が開きます。
解析例1
例として、乳がんにおけるBRCA1遺伝子変異がmRNA発現全体にどのような影響を与えるかを調べることができます。具体的にはStep1で「TCGA_BRCA」を選択し、Step2で「TCGA_BRCA, Mutation, Gene」を選択します。次に、Step3 に「BRCA1」と入力し、Step4 でTCGA_BRCA、RNAseqを選択、Step5でT-testを選択してSubmitボタンをクリックします。
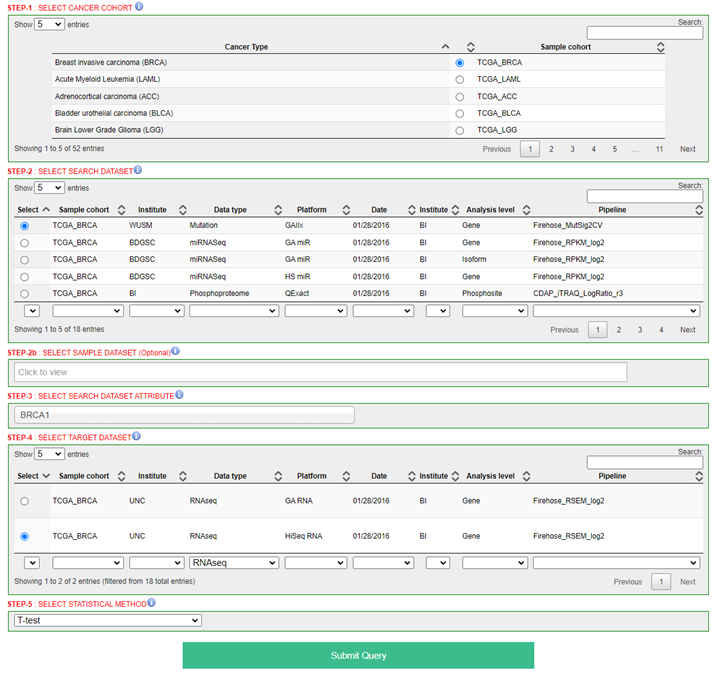
サブミットすると以下の画面が表示されます。
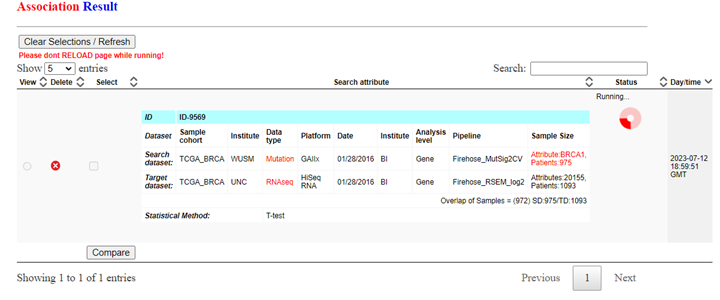
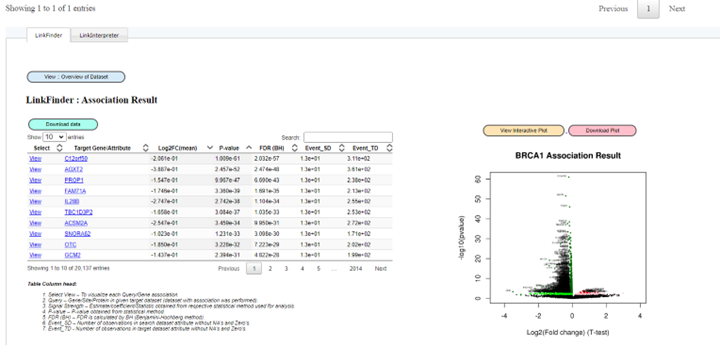
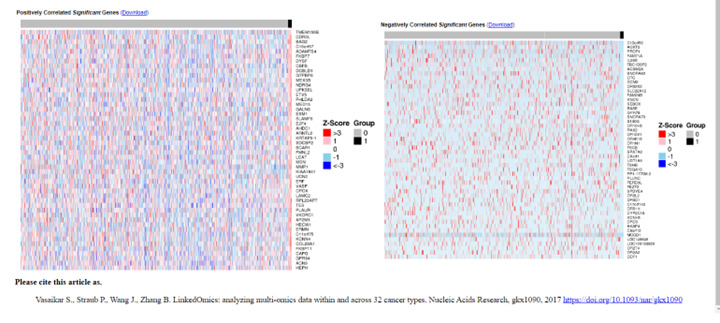
解析終了すると以下のような結果が表示されます。上段にはBRCA1変異vs野生型での発現差解析の結果がテーブルとVolcanoプロットで表示さています。下段のヒートマップはBRCA遺伝子発現量に対して、有意な正相関、負相関を示した遺伝子のヒートマップが表示されています。
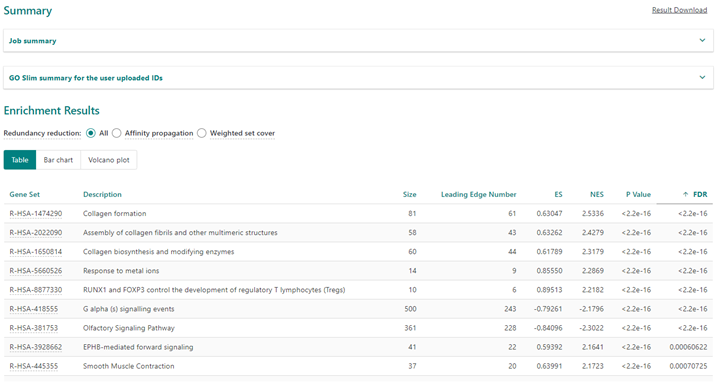
次にLinkInterpreterタブをクリックするとエンリッチメント解析ができます。遺伝子セットのみ情報で解析するOverrepresentative Enrichment Analysisと遺伝子名およびその発現値(正確には解析者に定義された群間での発現差。今回の例でいうとLog2 fold change valueのこと、GSEAと呼ばれている)が選択できます。今回はGSEAを選択し進めます。パスウェイデータベースとしてはGO、KEGG、Reactomeなどが選択できます。
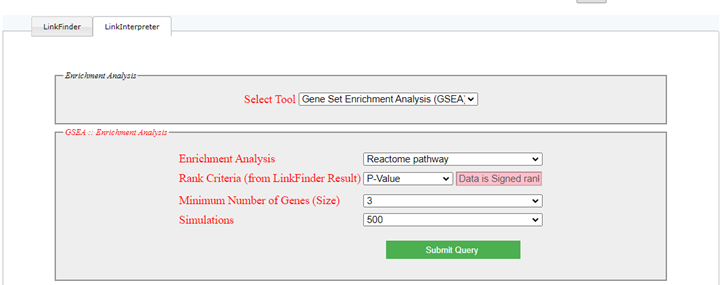
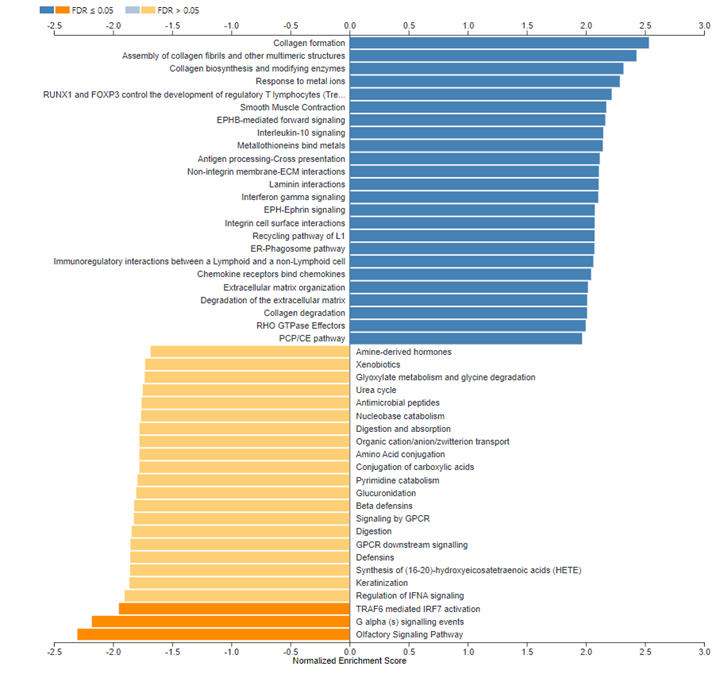
解析終了すると表、バーチャート、Volcanoプロットが自動的に生成され、ダウンロードできます。指定していなくてもGO_Slim 解析は実施されるようです。

解析例2
次に癌腫横断的な遺伝子変異の影響を調べたいときに役に立つのが、LinkCompareです。今回はBRCA1変異によるmRNA発現への影響を乳がん(BRCA)と膀胱がん(BLCA)調べ、癌腫にかかわらず共有されている変化を調べてみます。まず検索クエリ画面でBLCAのBRCA1変異を入力し、結果を入手します。
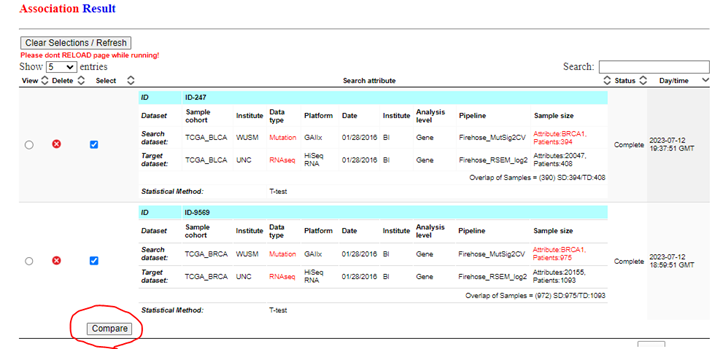
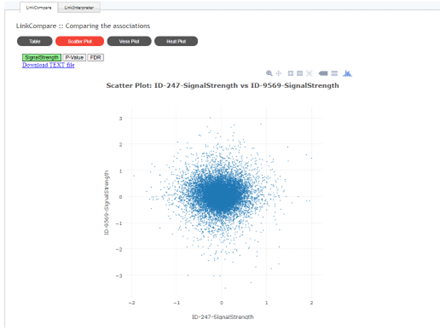
結果がそろったら、両方の結果を選択し、Compareをクリックすると表や散布図などの結果が表示されます。
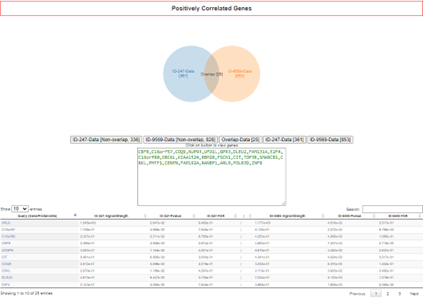
散布図やベン図でどのような共通因子があるのかがわかります。今回の結果では乳がんと膀胱がんでBRCA1変異の影響に関してシェアされた特徴がほぼないと解釈できると思います。
今回紹介した機能以外には、生存プロットやボックスプロットを書く機能のあるので、興味がある方はご自分で実施してみるとより理解が深まると思います。
今回も最後までお読みいただきありがとうございました。

